| X连锁鱼鳞病(X-linked ichthyosis,XLI)是由类固醇硫酸酯酶基因(STS)缺失或突变引起的皮肤角化障碍性疾病, 85% ~ 90%的 XLI病例为包括STS基因在内的Xp22.31片段缺失型,其余为STS单基因突变型。低深度全基因组测序拷贝数变异(CNV)检测技术(CNV-Seq)是近年来发展起来的快速、高效、准确的DNA片段缺失重复的新手段。本文探讨CNV-Seq在缺失型XLI基因诊断及产前诊断中的应用价值和意义。
低深度全基因组测序拷贝数变异检测技术对缺失型X连锁鱼鳞病的检测效力及意义的分析研究
白周现 陈晨 苏利沙 徐慧 王聪慧 时盼来 孔祥东
作者单位:郑州大学第一附属医院遗传与产前诊断中心 450000
【引用本文】白周现,陈晨,苏利沙,等. 低深度全基因组测序拷贝数变异检测技术对缺失型X连锁鱼鳞病的检测效力及意义的分析研究[J].中华皮肤科杂志,2019, 52(10): 736-742. doi:10.35541/cjd.20190197
本文第一作者:
白周现
【关键词】 鳞癣,X染色体连锁;DNA拷贝数变异;基因缺失;序列分析,DNA;产前诊断;Xp22.31缺失
收集2018年在郑州大学第一附属医院行CNV检测的3 616例案例资料及7例鱼鳞病患者或家系单基因检测资料。3 616例样本包括2 891例孕妇产前检测样本(主要为羊水,部分为胎儿绒毛,极少数为脐血样本)和725例其他受试者的外周血检测样本。分别提取羊水细胞和外周血等基因组DNA,应用CNV-Seq检测受试者DNA。定量PCR(qPCR)及单核苷酸多态性(SNP)-比较基因组杂交(CGH)微阵列法验证CNV-Seq检出的CNV。参考人群多态性数据库DGV、病例数据库DECIPHER、基因剂量效应数据库ClinGen、人类孟德尔遗传在线数据库OMIM等分析检出的CNV致病性。
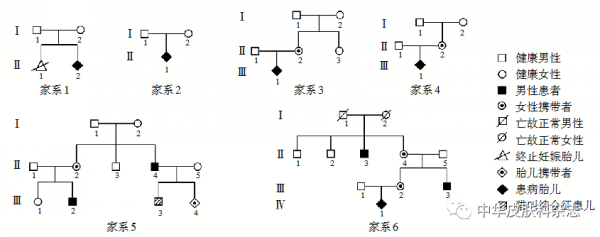
图1 6个X连锁鱼鳞病(XLI)家系谱图家系5中Ⅲ3为猫叫综合征患儿;家系1、2只追踪到2代人,受试者经随访否定鱼鳞病家族史
3 616例CNV检测案例中,6例孕妇产前检测样本存在Xp22.31缺失,包括5例男性胎儿和1例女性胎儿。检出的Xp22.31缺失片段覆盖XLI(主效基因STS)区域。经胎儿父母CNV验证,其中2例为自发突变,4例为父源或母源遗传。qPCR验证此6例胎儿,其中1例女性胎儿为STS基因完全杂合缺失携带者,5例男性胎儿STS基因完全缺失。SNP-CGH微阵列法验证1例女性胎儿杂合Xp22.31缺失携带者,结果与CNV检测结果一致。7例鱼鳞病患者进行鱼鳞病基因panel检测显示,丑角样鱼鳞病1例,寻常型鱼鳞病2例,XLI 3例,未找到致病基因突变1例。对其中2例缺失型XLI患者进行CNV检测显示存在Xp22.31缺失。此外,3 616例CNV测序案例中发现16例存在Xp22.31重复,但均为正常表型个体或胎儿。
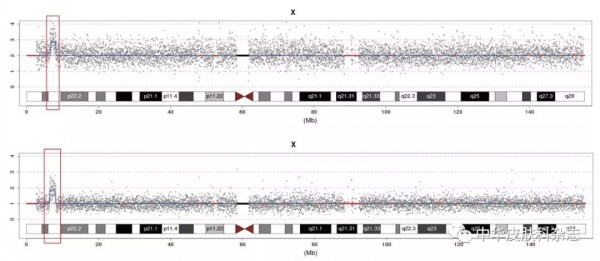
图3 拷贝数变异(CNV)检测显示部分受试者存在包含类固醇硫酸酯酶(STS)基因的Xp22.31片段重复 3A:女性受试者; 3B:男性受试者,红色方框内为拷贝数异常区域,其他区域拷贝数正常,显示Xp22.31片段重复
CNV-Seq检测技术稳定可靠,能够筛查全基因组范围内的CNVs,可用于缺失型XLI的基因诊断及产前诊断。包含STS基因的Xp22.31片段缺失可导致XLI,而含STS基因的Xp22.31片段重复为多态性变异可能性大。
|

